Introduction
L’analyse d’images microscopiques est au cœur de la recherche en sciences de la vie. Extraire automatiquement la forme et les contours des cellules est une tâche complexe et essentielle, notamment pour suivre la croissance cellulaire, détecter des anomalies ou encore analyser des données massives issues de la biologie médicale.
Cellpose-SAM est un modèle d’intelligence artificielle de nouvelle génération conçu pour relever ce défi. Développé par l’équipe MouseLand, il se distingue par sa capacité de généralisation “surhumaine” : il peut segmenter des cellules et des noyaux dans des conditions extrêmement variées, sans nécessiter un entraînement lourd ou spécifique à chaque laboratoire.
Qu’est-ce que Cellpose-SAM ?
Cellpose-SAM (Segment Anything Model) est un outil d’IA pour la segmentation d’images biologiques. Sa force repose sur sa robustesse et son adaptabilité :
- 📐 Travail en 2D et 3D : il fonctionne aussi bien sur des coupes microscopiques que sur des volumes 3D.
- 🌫️ Résistant au bruit et aux artefacts : bruit de type shot noise, flous isotropiques ou anisotropiques, sous-échantillonnage.
- 🔄 Flexible face aux canaux et contrastes : il s’adapte aux inversions de contraste, à l’ordre des canaux, et à la diversité des tailles cellulaires.
- 🛠️ Optimisation personnalisée : il peut être affiné (fine-tuned) sur vos propres données pour des résultats encore plus précis.
En résumé, Cellpose-SAM est pensé comme un outil universel pour accélérer les analyses biologiques, même dans des environnements d’imagerie difficiles.
Exemple d’image produite
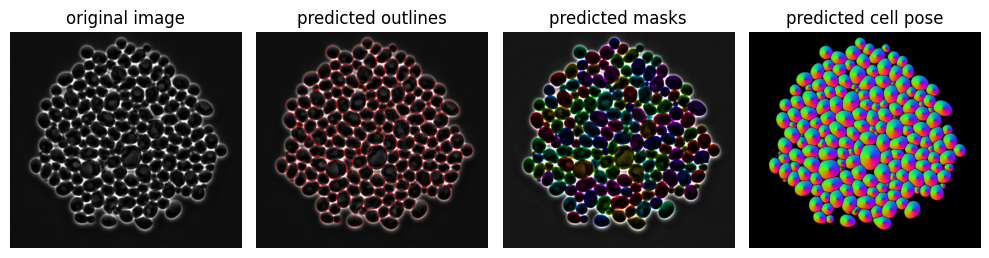
Afin d’illustrer concrètement le fonctionnement de Cellpose-SAM, les images ci-dessous montrent les différentes étapes du traitement. On commence par l’image originale, issue directement du microscope, qui représente les cellules sans aucune annotation. Le modèle génère ensuite les contours prédits, qui délimitent la forme de chaque cellule détectée. Ces contours sont transformés en masques prédits, où chaque cellule est colorée de manière distincte pour faciliter leur identification et leur comptage. Enfin, l’étape Cellpose prédit propose une visualisation avancée combinant les flux de segmentation et la forme globale des cellules, offrant une compréhension plus fine de la structure cellulaire.
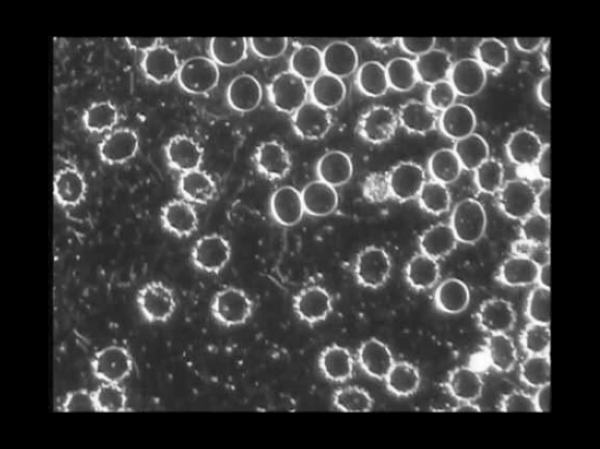
Image brute issue du microscope, avant tout traitement ou segmentation.
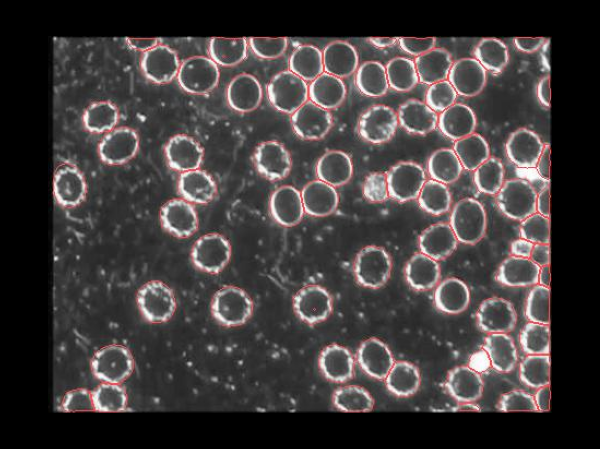
Les contours des cellules détectées par le modèle sont dessinés en surbrillance.
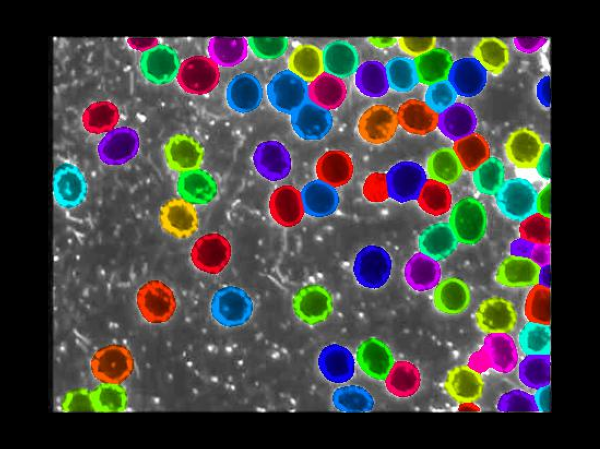
Chaque cellule est représentée par une zone colorée unique, permettant de les différencier et de les compter.

Visualisation avancée générée par Cellpose : flux de segmentation et formes cellulaires complètes.
Exemples supplémentaires
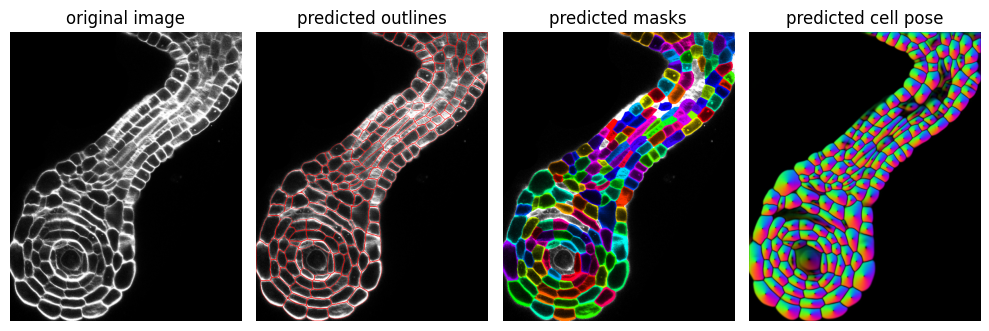
Limitations à connaître
Bien que puissant, Cellpose-SAM n’est pas sans limites :
- ⚡ L’utilisation en 3D et sur de grands volumes peut être coûteuse en ressources matérielles (GPU recommandé).
- 🎯 L’algorithme peut produire des erreurs de segmentation sur des images très atypiques si aucun ajustement n’est fait.
- 👩💻 Une certaine familiarité avec Python est nécessaire pour l’utiliser efficacement, bien que des interfaces conviviales existent.
Ces limites sont néanmoins compensées par la possibilité de réaliser un apprentissage interactif (human-in-the-loop), où l’utilisateur corrige progressivement les résultats pour améliorer le modèle.
Ressources pour apprendre et approfondir
- 📄 Article scientifique : Cellpose-SAM sur bioRxiv
- 🎥 Conférence introductive : Présentation sur YouTube
- 📚 Documentation officielle : Cellpose ReadTheDocs
- 💻 Code source : GitHub MouseLand
Pour toute question technique, vous pouvez ouvrir une “issue” sur GitHub.
Tester Cellpose-SAM avec Colab
Un projet Google Colab simplifié sera bientôt disponible pour tester Cellpose-SAM directement en ligne (aucune installation locale requise).
👉 [Lien à vers le notebook Colab]
Exécuter le code en local
Vous pouvez aussi installer l’outil en local :
pip install git+https://github.com/mouseland/cellpose.git
Exemple de code expliqué pas à pas
Voici un script minimaliste pour utiliser Cellpose-SAM sur une image. Chaque ligne est documentée pour les débutants. Vous devrez modifier la variable image_path et spécifier le chemin vers votre image à segmenter. Par la suite
# ------------------------------------------------------------
# Installation (à exécuter une fois, dans le terminal ou Colab)
# ------------------------------------------------------------
# pip install git+https://github.com/mouseland/cellpose.git
# pip install numpy
# pip install matplotlib
# ------------------------------------------------------------
# Importations
# ------------------------------------------------------------
# Fonctions principales de Cellpose (modèle, I/O, visualisation)
from cellpose import models, io, plot
# Manipulation efficace de tableaux numériques
import numpy as np
# Visualisation et sauvegarde de figures
import matplotlib.pyplot as plt
# Gestion des chemins et création de dossiers
import os
# ------------------------------------------------------------
# Prétraitement de l'image
# ------------------------------------------------------------
# Définition d'une fonction utilitaire pour charger et normaliser la forme de l'image
def preprocess_image(image_path: str) -> np.ndarray:
# """Docstring : prépare l'image pour Cellpose (HxWxC)"""
# Lecture du fichier image (PNG, TIFF, JPG, etc.)
img = io.imread(image_path)
# Suppression des dimensions "batch/page" de taille 1 au début, ex: (1,H,W,3) → (H,W,3)
while img.ndim >= 3 and img.shape[0] == 1 and img.ndim in (3, 4):
img = img[0]
# Conversion channels-first (C,H,W) vers channels-last (H,W,C) si nécessaire
if img.ndim == 3 and img.shape[0] in (1, 3) and img.shape[-1] not in (1, 3):
img = np.moveaxis(img, 0, -1)
# Ajout d'un canal si l'image est 2D (gris), ex: (H,W) → (H,W,1)
if img.ndim == 2:
img = img[..., None]
# Retourne l'image prête pour le modèle (HxWxC)
return img
# ------------------------------------------------------------
# Exécution du modèle Cellpose
# ------------------------------------------------------------
# Définition d'une fonction pour lancer l'inférence et obtenir masques/flux/styles
def run_cellpose(
img: np.ndarray,
gpu: bool = False,
channels = [0, 0],
flow_threshold: float = 0.4,
cellprob_threshold: float = 0.0,
):
# """Docstring : exécute Cellpose sur une image unique"""
# Création du modèle (activer gpu=True si CUDA disponible pour accélérer)
model = models.CellposeModel(gpu=gpu)
# Exécution de l'évaluation (segmentation) sur l'image
masks, flows, styles = model.eval(
img,
channels=channels,
flow_threshold=flow_threshold,
cellprob_threshold=cellprob_threshold,
)
# Retour des sorties principales : masques, champs de flux, styles
return masks, flows, styles
# ------------------------------------------------------------
# Visualisation et sauvegarde des résultats
# ------------------------------------------------------------
# Définition d'une fonction pour sauvegarder les masques et l'overlay PNG
def visualize_and_save(
img: np.ndarray,
masks: np.ndarray,
flows,
out_dir: str = "outputs",
mask_name: str = "mask.tif",
overlay_name: str = "overlay.png",
dpi: int = 150,
):
# Création du dossier de sortie s'il n'existe pas
os.makedirs(out_dir, exist_ok=True)
# Chemin complet pour le fichier de masque (TIFF, entier non signé 16 bits)
mask_path = os.path.join(out_dir, mask_name)
# Chemin complet pour l'image de superposition (PNG)
overlay_path = os.path.join(out_dir, overlay_name)
# Sauvegarde du masque de segmentation (conversion en uint16)
io.imsave(mask_path, masks.astype(np.uint16))
# Création d'une figure pour afficher image + contours/flux
fig = plt.figure(figsize=(8, 8))
# Affichage de la segmentation (utilise flows[0] pour une seule image)
plot.show_segmentation(fig, img, masks, flows[0])
# Ajustement automatique des marges
plt.tight_layout()
# Sauvegarde de la figure en PNG avec la résolution choisie
plt.savefig(overlay_path, dpi=dpi)
# Fermeture de la figure pour libérer la mémoire
plt.close()
# Affichage d'un petit récapitulatif en console
print(f"✅ Fichiers enregistrés : {mask_path} et {overlay_path}")
# ------------------------------------------------------------
# Fonction utilitaire : exécuter un pipeline complet sur un chemin d'image
# ------------------------------------------------------------
# Définition d'une fonction "pipeline" qui enchaîne prétraitement → modèle → sauvegarde
def process_image(
image_path: str,
gpu: bool = False,
channels = [0, 0],
flow_threshold: float = 0.4,
cellprob_threshold: float = 0.0,
out_dir: str = "outputs",
mask_name: str = "mask.tif",
overlay_name: str = "overlay.png",
):
# Prétraitement : lecture et mise en forme de l'image
img = preprocess_image(image_path)
# Inférence : segmentation avec Cellpose
masks, flows, styles = run_cellpose(
img=img,
gpu=gpu,
channels=channels,
flow_threshold=flow_threshold,
cellprob_threshold=cellprob_threshold,
)
# Sauvegardes : masque TIFF + overlay PNG
visualize_and_save(
img=img,
masks=masks,
flows=flows,
out_dir=out_dir,
mask_name=mask_name,
overlay_name=overlay_name,
)
# Retourne les objets si besoin d'un post-traitement ultérieur
return img, masks, flows, styles
# ------------------------------------------------------------
# Point d'entrée (exemple minimal)
# ------------------------------------------------------------
# Protection standard pour exécuter le script directement
if __name__ == "__main__":
# Spécification du chemin vers l'image d'exemple (à adapter à votre fichier)
image_path = "cellpose_img/003_img.png"
# Lancement du pipeline complet (GPU désactivé par défaut)
img, masks, flows, styles = process_image(
image_path=image_path,
gpu=False, # Mettre True si votre machine dispose de CUDA
channels=[0, 0], # Bon défaut pour brightfield/RGB (intensité en canal 0)
flow_threshold=0.4, # Seuil de qualité des contours (à ajuster au besoin)
cellprob_threshold=0.0, # Seuil de probabilité de cellule (à ajuster au besoin)
out_dir="outputs", # Dossier de sortie pour les résultats
mask_name="leaf_mask.tif",
overlay_name="leaf_overlay.png",
)
# Message final pour confirmer la fin du traitement
print("🎉 Traitement terminé.")
Conclusion
Cellpose-SAM ouvre de nouvelles perspectives en analyse automatisée d’images biologiques. Pour les étudiants, il s’agit d’un outil concret qui illustre la puissance de l’intelligence artificielle appliquée à la recherche scientifique. Son génie est de proposer une solution généraliste, flexible et accessible, tout en laissant la possibilité de l’adapter à ses propres données.
👉 Dans un prochain billet, nous partagerons un notebook Colab prêt-à-l’emploi pour tester Cellpose-SAM directement dans votre navigateur.
