Dans l’ère actuelle où les données de biologie moléculaire prolifèrent, en particulier les données de transcription au niveau cellulaire unique (scRNA-seq), un nouveau paradigme émerge : celui de considérer une cellule comme une phrase. Le projet Cell2Sentence (C2S) propose justement de représenter les profils d’expression génique de chaque cellule comme une « cell sentence », c’est-à-dire une séquence de noms de gènes triés par ordre décroissant d’expression, permettant d’exploiter les modèles de langage (LLM) dans le domaine de la biologie.
L’idée est à la fois simple et ambitieuse : si les grands modèles de langage (comme GPT‑4, Pythia, Gemma…) peuvent « lire » et « écrire » des phrases humaines, alors pourquoi ne pourraient-ils pas « lire » et « écrire » des cellules, en les convertissant en phrases de gènes ? Le pari est que la « langue des cellules » peut être apprise par ces modèles, d’où le sous-titre « Teaching Large Language Models the Language of Biology ». (Proceedings of Machine Learning Research)
Table des matières
- Contexte : pourquoi cette approche ?
- Principes techniques de C2S : transformation, modèles, tâches.
- Applications et résultats marquants.
- Ressources, code, utilisation pratique.
- Enjeux, limites et perspectives.
- Références.
1. Contexte : pourquoi convertir des cellules en phrases ?
Le défi des données de transcription cellulaire
La technologie de séquençage d’ARN à l’échelle d’une seule cellule (scRNA-seq) permet de mesurer l’expression de milliers de gènes dans chaque cellule, offrant une vision très fine de la diversité et de l’état cellulaire dans un tissu, un organe ou un état pathologique. Toutefois :
- Les matrices d’expression sont très hautes dimensions (ex : des milliers de gènes × des dizaines de milliers voire millions de cellules).
- L’interprétation et l’analyse nécessitent des pipelines spécialisés (normalisation, réduction de dimension, clustering, annotation, etc.).
- Il existe peu d’approches « universelles » permettant de poser des questions en langage naturel comme : « Que fait cette cellule ? Quel est son type ? Comment va-t-elle réagir à un traitement ? »
L’analogie avec le langage
Les grands modèles de langage ont montré une étonnante capacité à modéliser du texte, à générer des phrases cohérentes, à répondre à des questions, etc. L’idée de C2S est de « faire parler » les cellules, non pas au sens littéral, mais de les représenter sous forme de phrases que les LLM peuvent ingérer. Cette représentation permet de tirer parti :
- des architectures et de l’écosystème existants pour les LLM,
- de la richesse des métadonnées biologiques (types cellulaires, tissus, pathologies) souvent exprimées en texte,
- de la possibilité de poser des requêtes en langage naturel à des données cellulaires.
En d’autres termes, C2S cherche à établir un « langage de la biologie cellulaire ».
2. Principes techniques de C2S
La transformation « cell sentence »
Au cœur de C2S se trouve cette transformation :
- Pour chaque cellule issue d’un jeu de données scRNA-seq, on extrait son vecteur d’expression.
- On filtre/normalise selon les bonnes pratiques (ex : cellules avec trop peu/gêne trop peu exprimés, contrôle mitochondriale, etc.). (PMC)
- On ordonne les gènes selon leur niveau d’expression dans cette cellule, du plus exprimé au moins exprimé.
- On génère une séquence de noms de gènes, séparés par des espaces, c’est la « cell sentence ».
- Il est possible de conserver l’ordre comme seule information, car on montre que l’ordre (le rang) encode déjà beaucoup de l’information d’expression.
Fine-tuning de LLM pour la biologie
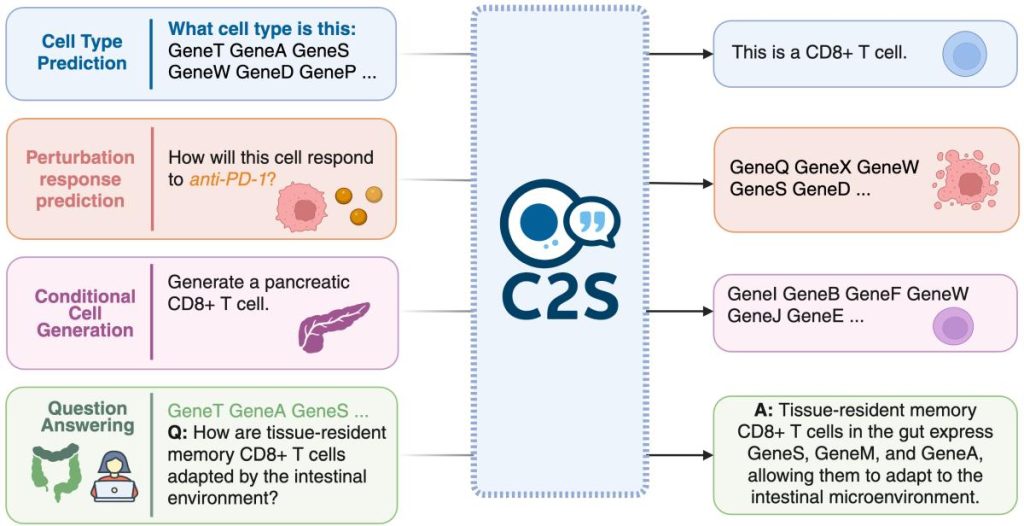
Une fois les cellules converties en phrases, on utilise un modèle de langage pré-entrainé (par ex GPT-4, Pythia, Gemma…) et on le fine-tune sur les « cell sentences ». On peut lui faire apprendre différentes tâches biologiques, par exemple :
- Génération de « cellules » (séquences de gènes) conditionnée sur un type cellulaire, un traitement, etc.
- Prédiction de l’étiquette cellulaire (type, tissu, condition) à partir de la « cell sentence ».
- Résumé en langage naturel d’un profil cellulaire ou d’un groupe de cellules : « cette cellule ressemble à un lymphocyte T activé exprimant X, Y, Z ».
Architecture & montée en échelle
Dans les travaux les plus récents, l’équipe du van Dijk Lab (Yale) et de Google Research/DeepMind ont proposé la version « C2S-Scale », évoluant vers des modèles de 27 milliards de paramètres. Ces modèles combinent les données transcriptomiques (sous forme de « cell sentences ») et du texte biologique/métadonnées afin d’apprendre un modèle « biologie + langue ». On y observe des lois d’échelle analogues à celles des LLM classiques : plus le modèle est grand, meilleures sont les performances sur les tâches biologiques. (Voir Google Research)
3. Applications & résultats marquants
Performances sur tâches de biologie cellulaire
Dans la publication initiale (ICML/MLResearch) : on montre qu’après fine-tuning avec C2S, un modèle GPT-2 est capable de :
- Générer des « cellules » (séquences de gènes) plausibles à partir d’une invite du type « cellule de type X ».
- Prédire correctement le type cellulaire à partir d’une « cell sentence ».
- Générer du texte (résumé logique, « abstract ») à partir d’un profil cellulaire : ce qui montre que le modèle a appris non seulement la structure « séquence de gènes », mais aussi des liens biologiques. biorxiv.org+1
Exemple concret : cellules « virtuelles » et simulation de perturbation
Grâce à la version C2S-Scale, les auteurs illustrent des usages très concrets :
- Générer des cellules représentatives de différents tissus ou états biologiques.
- Simuler l’effet d’une perturbation (ex : traitement médicamenteux, stimulation) en modifiant la phrase d’entrée et en générant une nouvelle phrase (« comment cette cellule changerait-elle ?»). Google Research+1
- Résumés automatiques de jeux de données entiers, annotation cellulaire automatique, etc.
Impact et cas d’usage
Cette approche ouvre des perspectives intéressantes :
- Faciliter l’annotation de types cellulaires dans des jeux de données scRNA-seq sans longue intervention manuelle.
- Permettre des interactions en langage naturel avec des données biologiques : « qu’est-ce que cette cellule fait ? », « quelle est sa réponse probable à ce traitement ? ».
- Créer des « modèles virtuels » de cellules ou de tissus, potentiellement utiles pour la découverte de thérapies ou l’expérimentation in silico.
4. Ressources, usage pratique et code
Code et documentation
- Le code source du projet est disponible sur GitHub : vandijklab/cell2sentence
- La documentation complète est hébergée sur Read the Docs : vandijklab-cell2sentence.readthedocs.io
- Le papier et les prépublications sont accessibles : voir notamment : Levine et al., Cell2Sentence: Teaching Large Language Models the Language of Biology, doi:10.1101/2023.09.11.557287. Centre National de Données Génomiques
Installation rapide
Extrait de la documentation :
git clone https://github.com/vandijklab/cell2sentence.git
conda create -n cell2sentence python=3.8
conda activate cell2sentence
make install
# ou pip install cell2sentence==1.1.0
Optionnellement, installation de flash-attn pour accélérer l’inférence. GitHub
Utilisation typique
Les tutoriels proposés incluent :
- Préparation des données (chargement, filtrage scRNA-seq)
- Transformation en « cell sentences »
- Fine-tuning d’un modèle C2S pour tâche spécifique (ex : prédiction de type cellulaire)
- Génération de cellules ou de résumés textuels
- Annotation automatique de cellules ou clusters (GitHub)
Modèles pré-entraînés (Model Zoo)
Le dépôt mentionne des modèles prêts à l’usage :
- C2S-Pythia-410M (pour prédiction de type cellulaire, génération conditionnée) GitHub
- Modèles plus grands (C2S-Scale) basés sur Gemma-2 ou autres architectures jusqu’à 27 milliards de paramètres. GitHub
- Modèle à télécharger sur Hugginface (55 Go)
5. Enjeux, limites et perspectives
Forces
- La transformation « cell sentence » est élégante : simple à comprendre, implémenter, et elle conserve une bonne part de l’information d’expression.
- Elle permet de tirer parti des vastes progrès des LLM dans un domaine jusque-là différent (biologie cellulaire).
- Offre une interface plus naturelle (texte) pour interagir avec des données biologiques.
- Le code est open-source, ce qui facilite l’adoption, l’extension et la validation.
Limites
- Bien que l’ordre de gènes encode beaucoup d’information, il y a une perte : les valeurs absolues d’expression, la dynamique temporelle, la spatialisation cellulaire, etc., ne sont pas toutes capturées.
- Le modèle dépend de la qualité des données d’entrée (prétraitement, annotations, normalisation).
- Les modèles de grande taille (ex : 27B paramètres) nécessitent des ressources computationnelles importantes, ce qui limite leur usage à certaines structures.
- L’interprétation biologique des résultats générés reste délicate : générer une « cell sentence » plausible ne garantit pas qu’elle soit biologiquement recréable ou validable.
- Comme pour tout modèle puissant, il y a un risque d’hallucination ou de sortie peu fiable si la requête s’éloigne du domaine d’entraînement.
Perspectives
- Extension à d’autres modalités : multi-omics (ARN, protéine, épigénétique), spatial transcriptomics.
- Intégration d’un raisonnement causal : simuler des perturbations, des réseaux de régulation, etc.
- Interface conversationnelle : demander à un modèle C2S « que ferait cette cellule si on inhibait ce gène ? » ou « dans quel état est ce groupe de cellules ? ».
- Validation expérimentale accrue : générer des hypothèses biologiques à tester en laboratoire.
- Réduction des besoins en ressources : modèles plus légers, fine-tuning efficace, quantification, déploiement sur matériel plus modeste.
6. Références
- Levine D., Rizvi S. A., Lévy S., Pallikkavaliyaveetil N., Zhang D., Chen X., Ghadermarzi S., Wu R., Zheng Z., Vrkic I., et al. Cell2Sentence: Teaching Large Language Models the Language of Biology. Proceedings of the 41ᵗʰ International Conference on Machine Learning (ICML) / Proceedings of Machine Learning Research 235:27299-27325, 2024. Proceedings of Machine Learning Research
- Levine D., Rizvi S. A., Lévy S., et al. Cell2Sentence: Teaching Large Language Models the Language of Biology. bioRxiv preprint, doi:10.1101/2023.09.11.557287, 2023. biorxiv.org+1
- GitHub repository : vandijklab/cell2sentence (https://github.com/vandijklab/cell2sentence) GitHub
- Documentation Read the Docs : Cell2Sentence – Single-cell Analysis with LLMs (https://vandijklab-cell2sentence.readthedocs.io) Cell2Sentence
- Blog post (Google Research/DeepMind & van Dijk Lab) : How a Gemma model helped discover a new potential cancer therapy pathway (15 octobre 2025) – décrivant C2S-Scale et ses usages. blog.google